來源:醫工學人
前言
從蛋白質工程、3D打印,到深度偽造介質的檢測,以下是【自然】期刊在未來一年將關註的七個技術領域。

插圖:雙胞胎計畫
Nature | Technology Feature
一、用於蛋白質設計的深度學習
二十年前,西雅圖華盛頓大學的大衛·貝克(David Baker)和他的同事們取得了一項裏程碑式的壯舉:他們使用計算工具從零開始設計了一種全新的蛋白質。「Top7」蛋白如預測的那樣折疊,但它是惰性的:它無法執行有意義的生物學功能。如今,蛋白質設計方法已經成熟,發展為一種實用工具,用於生成客製酶和其他蛋白質。「這極大地增強了科研效率」,華盛頓大學的生物化學家尼爾·金(Neil King)說,他與貝克的團隊合作設計了基於蛋白質的疫苗和藥物遞送載體。「一年半前不可能完成的事情——現在你只需要去做。」
這些進展很大程度上歸因於將蛋白質序列與結構聯系起來的日益龐大的數據集。但是,復雜的深度學習方法(人工智慧的一種形式)也是必不可少的。
「基於序列」的策略使用大型語言模型 (LLM),這些模型為 ChatGPT 等工具提供支持(參考:'ChatGPT?或許明年')。透過將蛋白質序列視為包含多肽「單詞」的文件,這些演算法可以辨別真實世界蛋白質架構劇本背後的模式。「他們真的學會了隱藏的語法,」西班牙巴塞隆納分子生物學研究所的蛋白質生物化學家Noelia Ferruz說。2022 年,她的團隊開發了一種名為 ProtGPT2 的演算法,該演算法始終如一地提出合成蛋白質,這些蛋白質在實驗室生產時可以穩定折疊[1]。Ferruz共同開發的另一種工具ZymCTRL利用序列和功能數據來設計天然存在的酶家族的成員[2]。
基於序列的方法可以建立在現有蛋白質特征的基礎上並對其進行調整以形成新的框架,但它們對於結構元件或特征的客製設計效果較差,例如以可預測的方式結合特定靶標的能力。「基於結構」的方法更適合這一點,2023 年這種型別的蛋白質設計演算法也取得了顯著進展。其中一些最復雜的模型使用「擴散」模型,這也是影像生成工具(如DALL-E)的基礎。這些演算法最初經過訓練,可以從大量真實結構中去除電腦生成的雜訊;透過學習區分現實的結構元素和雜訊,他們獲得了形成生物學上合理的使用者定義結構的能力。
RFdiffusion 軟體[3]是由 Baker 的實驗室和麻薩諸塞州薩默維爾的 Generate Biomedicines 開發的色度工具[4],他們利用這一工具取得了顯著的設計效果。例如,Baker 的團隊正在使用 RFdiffusion 來設計新型蛋白質,「這些蛋白質可以與感興趣的靶標形成緊密的界面,從而產生「完全符合表面」的設計,」Baker 說。RFdiffusion 的更新的「全原子」叠代[5]允許設計人員圍繞非蛋白質靶標(如DNA、小分子甚至金屬離子)計算形狀蛋白質。由此產生的多功能性蛋白為工程酶、轉錄調節因子、功能性生物材料等蛋白質設計開辟了新的視野。
ChatGPT?或許明年
讀者可能會在23年的技術中發現一個值得關註的主題:深度學習的巨大影響。但有一個工具沒有進入最終階段:大肆宣傳的人工智慧(AI)驅動的聊天機器人。ChatGPT 及其同類產品似乎有望成為許多研究人員日常生活的一部份,並被視為 2023 年【自然】雜誌 10 大綜述的一部份(go.nature.com/3trp7rg)。9 月份【自然】雜誌中一項調查( go.nature.com/45232vd)的受訪者認為 ChatGPT 是最有用的基於人工智慧的工具,並對其在編碼、文獻綜述和管理任務方面的潛力充滿熱情。
從公平的角度來看,這些工具也被證明很有價值,可以幫助那些英語不是第一語言的人完善他們的論文,從而簡化他們的出版及職業發展之路。然而,其中許多套用代表了節省勞動力的收益,而不是研究過程的轉變。此外,ChatGPT 持續釋出誤導性或捏造的回復是超過三分之二受訪者的主要擔憂。雖然值得監測,但這些工具需要時間來發展成熟並確立它們在科學界的更廣泛作用。
二、Deepfake檢測
公開可用的生成式 AI 演算法的爆炸式增長使得合成令人信服但完全是人工的影像、音訊和視訊變得簡單。但結果可能會讓人擔心,隨著持續的地緣政治沖突和美國總統大選的臨近,媒體操縱的機會很多。
紐約水牛城大學(University at Buffalo)的電腦科學家呂思維(Siwei Lyu)說,他見過許多人工智慧生成的與以色列-哈馬斯沖突有關的「深度偽造」影像和音訊。這只是一場高風險的貓捉老鼠遊戲的最新一輪,在這場遊戲中,人工智慧使用者制作欺騙性內容,而呂和其他媒體取證專家則致力於檢測和攔截它。
一種解決方案是讓生成式 AI 開發人員在模型的輸出中嵌入隱藏訊號,從而生成浮水印。其他策略側重於內容本身。例如,「一些視訊將一個公眾人物的面部特征替換為另一個公眾人物的面部特征,而新的演算法可以在替換特征的邊界處辨識偽影,」Lyu說。一個人外耳的獨特褶皺也可以揭示臉部和頭部之間的不匹配,而牙齒的不規則性可以揭示經過編輯的對口型視訊,其中一個人的嘴巴被數位操縱以說出受試者沒有說的話。人工智慧生成的照片也是一個棘手的挑戰,也是一個移動的目標。2019 年,義大利拿坡里費德裏科二世大學的媒體取證專家 Luisa Verdoliva 幫助開發了 FaceForensics++,這是一種用於發現被幾個廣泛使用的軟體包操縱的人臉的工具[6]。但影像取證方法是特定於主題和軟體的,泛化是一個挑戰。「你不能有一個單一的通用檢測器——這非常困難,」她說。
然後是實施的挑戰。美國國防高等研究計劃署的語意取證 (SemaFor) 計劃開發了一個用於深度偽造分析的有用工具箱,但正如【自然】雜誌報道的那樣( Nature 621, 676–679; 2023),主流社交媒體網站並沒有經常使用它。擴大對此類工具的存取可能有助於促進使用,為此,Lyu 的團隊開發了 DeepFake-O-Meter[7],一個集中的公共演算法儲存庫,可以從不同角度分析視訊內容以嗅出深度偽造內容。這些資源將有所幫助,但與人工智慧產生的錯誤資訊的鬥爭可能會在未來幾年持續下去。
三、大片段DNA插入
2023 年底,美國和英國監管機構批準了第一個基於 CRISPR 的基因編輯療法,用於治療鐮狀細胞病和輸血依賴性地中海貧血β——這是基因組編輯作為臨床工具的重大勝利。
CRISPR 及其衍生物使用短可編程 RNA 將 DNA 切割酶(如 Cas9)引導至特定的基因組位點。它們在實驗室中通常用於禁用有缺陷的基因並引入小的序列變化。精確和可編程地插入跨越數千個核苷酸的較大DNA序列是很困難的,但新興的解決方案可以讓科學家替換有缺陷基因的關鍵片段或插入功能齊全的基因序列。加州史丹佛大學的分子遺傳學家Le Cong和他的同事們正在探索單鏈退火蛋白(SSAP)——介導DNA重組的病毒衍生分子。當與禁用Cas9的DNA切片功能的CRISPR-Cas系統結合使用時,這些SSAP允許將多達2千堿基的DNA精確靶向插入人類基因組中。
其他方法利用一種稱為質數編輯的基於CRISPR的方法引入短的「著陸墊」序列,這些序列選擇性地募集酶,而酶又可以將大的DNA片段精確地拼接到基因組中。例如,2022 年,劍橋麻省理工學院的基因組工程師 Omar Abudayyeh 和 Jonathan Gootenberg 及其同事首次描述了透過位點特異性靶向元件 (PASTE) 進行可編程添加,這種方法可以精確插入多達 36 千堿基的 DNA[8]。Cong說,PASTE在培養的、患者來源的細胞的離體修飾方面特別有前景,並且潛在的初免編輯技術已經進入了臨床研究的軌域。但對於人體細胞的體內修飾,SSAP可能提供更緊湊的解決方案:體積較大的PASTE機器需要三個獨立的病毒載體進行遞送,這可能會降低相對於雙組分SSAP系統的編輯效率。也就是說,即使是相對低效的基因替代策略也足以減輕許多遺傳疾病的影響。
這些方法不僅與人類健康有關。由北京中國科學院的Caixia Gao領導的研究人員開發了PrimeRoot,這是一種使用Prime Editing引入特定靶位點的方法,酶可以使用這些靶位點在水稻和玉米中插入多達20千個堿基的DNA[9]。Gao認為,該技術可用於賦予作物抗病性和抗病原體性,繼續推動基於CRISPR的植物基因組工程的創新浪潮。「我相信這項技術可以套用於任何植物物種,」她說。
四、腦機介面
帕特·貝內特(Pat Bennett)的語速比一般人慢,有時可能會用錯詞。但鑒於其罹患運動神經元疾病,也稱為肌萎縮側索硬化癥,以前的她甚至無法用語言表達自己,現在這是一項了不起的成就。
貝內特的康復得益於史丹佛大學神經科學家法蘭西斯·威利特(Francis Willett)及其在美國BrainGate聯盟的同事開發的復雜腦機介面(BCI)裝置[10]。威利特和他的同事在貝內特的大腦中植入電極來跟蹤神經元活動,然後訓練深度學習演算法將這些訊號轉化為語音。經過幾周的訓練,Bennett 每分鐘能夠從125,000個單詞的詞匯量中說出多達 62 個單詞——是普通英語使用者詞匯量的兩倍多。「他們交流的速度真的令人印象深刻,」在賓夕法尼亞州匹茲堡大學開發BCI技術的生物工程師Jennifer Collinger說。

腦機介面技術使帕特·貝內特(坐著)恢復了她的語言能力。圖片來源:Steve Fisch/史丹佛醫學
BrainGate的試驗只是過去幾年的幾項研究之一,這些研究證明了BCI技術如何幫助患有嚴重神經損傷的人重新獲得失去的技能並實作更大的獨立性。其中一些進展源於各種神經系統疾病患者大腦中功能性神經解剖學知識的穩步積累,羅德島州普羅維登斯布朗大學的神經學家、BrainGate聯盟主任Leigh Hochberg說。但他補充說,透過機器學習驅動的分析方法,這些知識已經大大放大,這些方法揭示了如何更好地放置電極並解密它們拾取的訊號。
研究人員還在套用基於人工智慧的語言模型來加速對患者試圖交流的內容的解釋——本質上是大腦的「自動完成」。這是威利特研究的一個核心組成部份,也是另一個[11]來自加州大學舊金山分校神經外科醫生愛德華·張(Edward Chang)領導的團隊。在這項工作中,腦機介面神經義肢允許一名因中風而無法說話的女性以每分鐘78個單詞的速度進行交流——大約是英語平均速度的一半,但比該女性以前使用的語音輔助裝置快五倍多。該領域在其他方面也取得了進展,2021 年,匹茲堡大學的 Collinger 和生物醫學工程師 Robert Gaunt 將電極植入一名四肢癱瘓的人的運動和軀體感覺皮層中,以提供對機械臂的快速精確控制以及觸覺反饋[12]。此外,BrainGate 和荷蘭烏特勒支UMC 研究人員的獨立臨床研究正在進行中,以及位於紐約布魯庫林的 BCI 公司 Synchron 的一項試驗,以測試一種允許癱瘓的人控制電腦的系統——這是第一個由行業贊助的 BCI 裝置試驗。
作為一名重癥監護專家,Hochberg 渴望將這些技術提供給殘疾最嚴重的患者。但隨著腦機介面能力的發展,他看到了治療中度認知障礙以及情緒障礙等心理健康狀況的潛力。「由腦機介面提供的閉環神經調控系統可能對很多人產生巨大的幫助,」他說。
五、超分辨技術
Stefan Hell、Eric Betzig 和 William Moerner 因突破限制光學顯微鏡空間分辨率的「繞射極限」而獲得 2014 年諾貝爾化學獎。由此產生的細節水平——在數十奈米的量級——開啟了廣泛的分子尺度成像實驗。盡管如此,一些研究人員仍然渴望更好,而且他們正在迅速取得進展。「我們真的在努力縮小從超分辨率顯微鏡到冷凍電子顯微鏡等結構生物學技術的差距,」德國普蘭埃格馬克斯普朗克生物化學研究所的奈米技術研究員Ralf Jungmann說,他指的是一種可以以原子級分辨率重建蛋白質結構的方法。
2022 年底,哥廷根馬克斯·普朗克多學科科學研究所 Hell 及其團隊領導的研究人員使用一種名為 MINSTED 的方法首次涉足這一領域,該方法可以使用專門的光學顯微鏡以 2.3 萬億的精度(大約四分之一奈米)解析單個熒光標記[13]。
較新的方法可提供與傳統顯微鏡相當的分辨率。例如,Jungmann 和他的團隊在 2023 年描述了一種方法,其中單個分子用不同的 DNA 鏈標記[14]。然後用染料標記的互補DNA鏈檢測這些分子,這些DNA鏈瞬時但重復地與相應的靶標結合,從而可以區分單個熒光「閃爍」點,如果同時成像,這些點會模糊成一個斑點。這種透過順序成像 (RESI) 方法增強的分辨率可以解析 DNA 鏈上的單個堿基對,從而使用標準熒光顯微鏡證明其分辨率為 ångström 尺度。
由德國哥廷根大學醫學中心的神經科學家Ali Shaib和Silvio Rizzoli領導的團隊開發的一步奈米級擴充套件(ONE)顯微鏡方法並沒有完全達到這種分辨率水平。然而,ONE顯微鏡提供了一個前所未有的機會,可以直接對單個蛋白質和多蛋白質復合物的精細結構細節進行成像,無論是在分離中還是在細胞中[15]。

一種稱為RESI的成像形式可以對DNA中的單個堿基對進行成像。圖片來源:Max Iglesias,馬克斯·普朗克生物化學研究所
ONE是一種基於擴增顯微鏡的方法,涉及將樣品中的蛋白質與水凝膠基質進行化學偶聯,將蛋白質分解,然後讓水凝膠膨脹1000倍。片段在各個方向上均勻擴充套件,保留了蛋白質結構,使使用者能夠使用標準共聚焦顯微鏡解析相隔幾奈米的特征。「我們取抗體,將它們放入凝膠中,在擴增後標記它們,然後說,」哦,我們看到了Y形!「Rizzoli說,指的是蛋白質的特征形狀。
Rizzoli說,ONE顯微鏡可以提供對構象動態生物分子的見解,或者能夠從血液樣本中直觀診斷蛋白質錯誤折疊疾病,如帕金森病。Jungmann 同樣熱衷於 RESI 記錄疾病中單個蛋白質重組或對藥物治療的反應的潛力。甚至可以更緊密地放大。「也許這不是空間分辨率限制的終點,」Jungmann說。「它可能會變得更好。」
六、細胞圖譜
如果您正在尋找方便的咖啡館,Google 地圖可以找到附近的選項並告訴您如何到達那裏。在更復雜的人體景觀中導航沒有等價物,但各種細胞圖譜計劃的持續進展——由單細胞分析和「空間組學」方法的進步提供支持——可能很快就會提供生物學家渴望的組織尺度下的細胞圖譜。
這些計劃中規模最大,也許也是最雄心勃勃的,是人類細胞圖譜(HCA)。該聯盟於2016年由英國欣克斯頓Wellcome Sanger研究所的細胞生物學家Sarah Teichmann和加利福尼亞州南舊金山生物技術公司Genentech的研究和早期開發負責人Aviv Regev發起。它包括近100個國家的約3,000名科學家,使用來自10,000名捐贈者的組織。但HCA也是細胞和分子圖譜交叉工作的更廣泛生態系的一部份。其中包括由美國國立衛生研究院資助的人類生物分子圖譜計劃(HuBMAP)和透過推進創新神經技術(BRAIN)倡議進行腦研究的細胞普查網路(BICCN),以及由華盛頓州西雅圖艾倫研究所資助的艾倫腦細胞圖譜。
史丹佛大學基因組學家、HuBMAP指導委員會前聯合主席麥可·斯奈德(Michael Snyder)表示,這些努力在一定程度上是由能夠在單細胞水平上解分碼子含量的分析工具的開發和快速商業化推動的。例如,Snyder的團隊經常使用位於加利福尼亞州普萊森頓的10X Genomics的Xenium平台進行空間轉錄組學分析。該平台每周可以一次調查 4 個組織樣本中大約 400 個基因的表達。基於多重抗體的方法,例如位於麻薩諸塞州馬爾堡的Akoya Biosciences的PhenoCycler平台,使該團隊能夠以單細胞分辨率跟蹤大量蛋白質,從而實作3D組織重建。其他「多組學」方法允許科學家同分時析同一細胞中的多個分子類別別,包括RNA的表達、染色質的結構和蛋白質的分布。
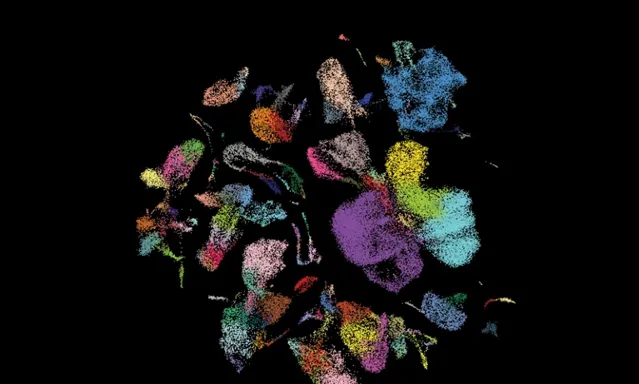
人肺的細胞圖譜描述了不同的細胞型別以及它們是如何調節的。圖片來源:Peng He
去年,數十項研究展示了使用這些技術生成器官特異性圖譜的進展。例如,今年6月,HCA釋出了對49個人類肺部數據集的綜合分析16.Teichmann 說:「擁有非常清晰的肺部地圖可以了解肺纖維化、不同腫瘤等疾病發生的變化,即使是 COVID-19。2023 年,【自然】雜誌釋出了一篇文章集(go.nature.com/3vbznk7),重點介紹了 HuBMAP 的進展,而【科學】雜誌則制作了一篇詳細介紹了 BICCN 工作的合集(go.nature.com/3nsf4ys)。
還有相當多的工作要做——Teichmann 估計 HCA 至少需要五年時間才能完成。但是,當它們到達時,生成的地圖將是無價的。例如,Teichmann預測使用圖譜數據來指導組織和細胞特異性藥物靶向,而Snyder則渴望了解細胞微環境如何告知癌癥和腸易激症候群等復雜疾病的風險和病因。「我們會在2024年解決這個問題嗎?我不這麽認為——這是一個多年的問題,「斯奈德說。「但這是整個領域的一大驅動力。
七、3D打印奈米材料
在奈米尺度上可能會發生很多奇怪而有趣的事情。這可能使材料科學預測變得困難,但這也意味著奈米級建築師可以制造出具有獨特特性的輕質材料,例如增加強度、與光或聲音的特定交互作用以及增強的催化或能量儲存能力。
有幾種策略可以精確地制作這種奈米材料,其中大多數使用雷射來誘導光敏材料的圖案化「光聚合」,並且在過去幾年中,科學家們在克服阻礙更廣泛采用這些方法的局限性方面取得了相當大的進展。

研究人員使用水凝膠制作了微尺度金屬結構。圖片來源:Max Saccone/Greer Lab
一是速度。亞特蘭大佐治亞理工學院的工程師Sourabh Saha表示,使用光聚合法組裝奈米結構的速度比其他奈米級3D打印方法快大約三個數量級。對於實驗室使用來說,這可能足夠好了,但對於大規模生產或工業過程來說,它太慢了。2019 年,Saha 和香港中文大學的機械工程師 Shih-Chi Chen 及其同事表明,他們可以透過使用圖案化的 2D 光片而不是傳統的脈沖雷射器來加速聚合[17]。「這將速率提高了一千倍,而且你仍然可以保持這些100奈米的特征,」Saha說。包括Chen在內的研究人員的後續工作已經確定了其他更快的奈米加工途徑[18]。
另一個挑戰是,並非所有材料都可以透過光聚合直接打印,例如金屬。但位於帕薩迪納的加州理工學院(California Institute of Technology)的材料科學家朱莉婭·格里耳(Julia Greer)開發了一種聰明的解決方法。2022 年,她和她的同事描述了一種將光聚合水凝膠用作微尺度樣版的方法;然後將它們註入金屬鹽,並以一種誘導金屬呈現樣版結構同時收縮的方式進行加工[19]。雖然該技術最初是為微尺度結構開發的,但Greer的團隊也將這種策略用於奈米制造,研究人員對從堅固的高熔點金屬和合金中制造功能性奈米結構的潛力充滿熱情。
最後一個障礙——經濟成本——可能是最難打破的。根據Saha的說法,許多光聚合方法中使用的基於脈沖雷射的系統成本高達50萬美元。但更便宜的替代品正在出現。例如,德國卡爾斯魯厄理工學院的物理學家馬丁·韋格納(Martin Wegener)和他的同事們已經探索了比標準脈沖雷射器更便宜、更緊湊、功耗更低的連續雷射器[20]。格里耳還成立了一家初創公司,將奈米結構金屬板的制造工藝商業化,該工藝可能適用於下一代防彈衣或飛機和其他車輛的超耐用和抗沖擊外層等套用。
doi: https://doi.org/10.1038/d41586-024-00173-x
參考資料
[1] Ferruz, N., Schmidt, S. & Höcker, B. Nature Commun. 13, 4348 (2022).
[2] Munsamy, G., Lindner, S., Lorenz, P. & Ferruz, N. ZymCTRL: A Conditional Language Model for the Controllable Generation of Artificial Enzymes (MLSB, 2022).
[3] Watson, J. L. et al. Nature 620, 1089–1100 (2023).
[4] Ingraham, J. B. et al. Nature 623, 1070–1078 (2023).
[5] Krishna, R. et al. Preprint at bioRxiv https://doi.org/10.1101/2023.10.09.561603 (2023).
[6] Rössler, A. et al. Preprint at https://arxiv.org/abs/1901.08971 (2019).
[7] Li, Y., Zhang, C., Sun, P., Qi, H. & Lyu, S. Preprint at https://arxiv.org/abs/2103.02018 (2021).
[8] Yarnall, M. T. N. et al. Nature Biotechnol. 41, 500–512 (2023).
[9] Sun, C. et al. Nature Biotechnol. https://doi.org/10.1038/s41587-023-01769-w (2023).
[10] Willett, F. R. et al. Nature 620, 1031–1036 (2023).
[11] Metzger, S. L. et al. Nature 620, 1037–1046 (2023).
[12] Sharlene, N. et al. Science 372, 831–836 (2021).
[13] Weber, M. et al. Nature Biotechnol. 41, 569–576 (2023).
[14] Reinhardt, S. C. M. et al. Nature 617, 711–716 (2023).
[15] Shaib, A. H. et al. Preprint at bioRxiv https://doi.org/10.1101/2022.08.03.502284 (2023).
[16] Sikkema, L. et al. Nature Med. 29, 1563–1577 (2023).
[17] Saha, S. K. et al. Science 366, 105–109 (2019).
[18] Ouyang, W. et al. Nature Commun. 14, 1716 (2023).
[19] Saccone, M. A. et al. Nature 612, 685–690 (2022).
[20] Hahn, V. et al. Nature Photon. 16, 784–791 (2022).
向下滑動檢視
推薦閱讀 點選標題可跳轉
1、
2、
3、










